迄今为止,临床宏基因组学的应用场景包括以下几个方面:
- 各种综合征和样本类型的传染病诊断
- 患病和健康状态下的微生物组特征分析
- 宿主转录学分析确定人类宿主对感染的反应特征
- 肿瘤领域的应用包括鉴定肿瘤相关病毒及鉴定基因组整合位点。
除传染病诊断外,mNGS其它领域的应用速度一直很慢,大多数应用尚未纳入常规临床实践。尽管如此,这些应用很可能在不久的将来改变诊断微生物学的领域。
分子诊断分析为诊断最常见的感染提供了一种快速经济有效的方法(通常小于2小时的周转时间)。然而,目前使用的几乎所有常规微生物试验一次只能检测一种或有限的病原体,或要求从临床样本中成功培养出微生物。与之相比,NGS的方法虽然耗费时间较长(在标准Illumina测序仪上需要耗费大于18个小时的时间),但是mNGS可以根据各个物种唯一的DNA或者RNA序列,一次性检测范围较广的病原菌,包括病毒、细菌、真菌或者寄生虫。 因此,NGS在诊断中的临床应用可能是在最难诊断的病例或免疫功能低下的患者中,在这些患者中潜在病原体的范围更大。最终,mNGS方法与其他多种检测方法相比更具成本竞争力,可以用作排除感染性病因的预先检测方法。当然,核酸类型的检测,无论是通过多重PCR 还是NGS,都不能单独证明一种是感染的病因,发现的结果必须在临床背景下进行解释。在临床样本中发现非典型或新型感染源时,需要进行后续的确认调查工作,例如组织活检样本的正交试验和血清试验,或者通过细胞培养以及动物模型的方法,以确定真正的致病菌。
在传染病诊断方面的应用。包括从主要临床样本中直接鉴定微生物(Aa部分);基于耐药基因鉴定的抗菌药物耐药性预测(Ab部分);检测种级或株级毒力决定因素,如分泌特定内毒素或外毒素(Ac部分);抗病毒药物耐药性预测(Ad部分)。如HIV-1所示,通过宏基因组下一代测序(mNGS)(部分Ad,图)从患者样本中恢复完整的病毒基因组有助于序列分析,以预测抗逆转录病毒药物的敏感性或耐药性(部分Ad,柱状图);分析菌株的易感谱(黑条)预测了对非核苷逆转录酶抑制剂(NNRTI)类药物的耐药性(用星号表示),而不是核苷逆转录酶抑制剂(NRTIs)或蛋白酶抑制剂(pi)
许多研究人员现在使用mNGS或16S rRNA基因靶向测序来深入描述微生物群落。越来越多的人意识到微生物组及其在急性和慢性疾病状态中的可能作用。 然而,基于微生物组的测试还没有被临床验证用于疾病的诊断或治疗,部分原因是由于对微生物组的复杂性及其在疾病发病机制中的作用的认识还不完全。
mNGS具有检测全面,准确率高,敏感度高和时间短的优势。鉴定样品中病原微生物时,mNGS可以补充或替代传统的生化、免疫和培养方法,更快更加准确地取得结果。更重要的是,传统方法在应对少见、罕见感染,新发性感染时常常束手无策,mNGS通过检测样品中全微生物物种,从而找出潜在的致病微生物,指导用药。
2.1在新发传染病疫情中的应用
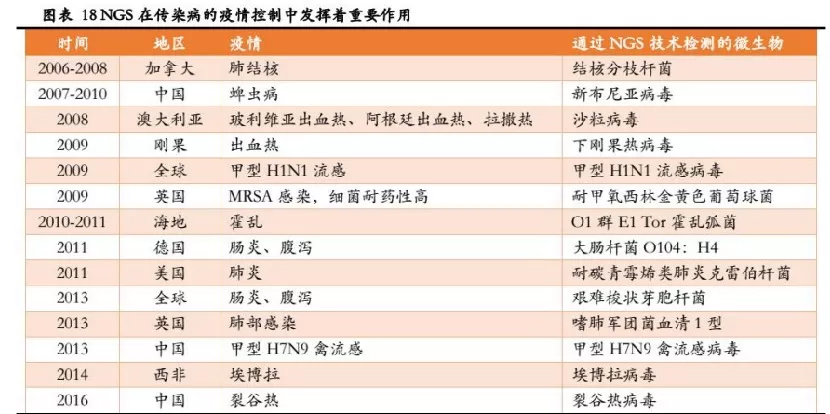
在新发传染病疫情的发现和控制中,mNGS发挥了越来越重要的作用。2011年德国产志贺毒素大肠杆菌(STEC)O104:H4暴发,没有相应诊断试剂及标准可用于该新亚型,给检测带来了极大的难度。在多个高通量测序系统合力开展了宏基因组测序分析后,成功完成了菌株的基因组序列的重建,迅速确认了致命德国肠出血性大肠杆菌O104:H4。至今,mNGS在多次疫情中检测出致病菌,起到了鉴别病菌、控制疫情的作用。
mNGS用于感染性疾病的诊断首次由加州大学Charles Y. Chiu团队于2014年报道,发表在《英格兰医学杂志》。1例14岁患者,头痛、发热反复住院8个月,在进行了38种不同的诊断试验(从血清学到培养和活检),仍然无法明确其致病病原体,并进入了昏迷状态。最终团队使用mNGS检测其脑脊液样品,确诊为钩端螺旋体感染。随即给予针对性的青霉素治疗,32天后患者痊愈出院。此后该项技术广泛用于多种感染性疾病的诊断。
据估计经过全面深入的传统检查后仍有30%~60%的脑炎病例无法明确其病因。使用mNGS检测可检查出所有微生物,帮助诊断病毒性(DNA病毒和RNA病毒)脑炎。2015年,德国报道了3例基底节脑炎患者,患者临床上以基底节病变为主,发病急骤,死亡率高,通过mNGS在脑内检测到松鼠博尔纳病毒,这是首次发现松鼠博尔纳病毒传染人类的证据。2015年,日本Sakiyama等报道了4例表现为脑脊髓膜炎的渔民,常规检查未能明确其致病病原体,通过mNGS检测发现其感染了以前从未曾报道的属于古细菌域的嗜盐菌(Halobacterium),在给予复方磺胺甲恶唑治疗后患者完全好转。上述报道均表明,mNGS检测在诊断罕见的、新发的或不典型的病原微生物(如钩端螺旋体、星状病毒、古细菌、博尔纳病毒)时发挥出了独特的优势,可以有效检测病原微生物并且指导用药。
在呼吸道感染中,除血液标本外,mNGS已用于多种呼吸道多种标本的检测,包括痰、咽拭子、胸水、支气管肺泡灌洗液、脓液及肺活检标本等。
报道一项关于mNGS技术应用于重症肺炎的病原体检测效果的回顾性研究,对照组及mNGS组共178例患者。对照组130例患者送检痰、血及支气管肺泡灌洗液标本做常规的微生物培养,根据培养结果调整抗菌药物使用;mNGS组48例患者加做mNGS,根据mNGS检测结果调整抗菌药物的使用。最后以28天,90天死亡率为指标,mNGS组均显著低于对照组,说明在mNGS结果的辅助下,患者的治愈率显著更高。胡必杰等报道了应用mNGS检测病原体的一项大样本回顾性研究分析,纳入可分析数据的511例患者。通过分析发现mNGS较传统培养方法敏感性提高近15%。
基于NGS方法优化的测序方法分别对94份重症病例和150份轻症病例临床样本进行了测序分析,共检出哺乳动物病毒科和植物病毒科共13种病毒。此外还检测出了多种肠道病毒不同型别之间、肠道病毒与非肠道病毒之间的混合感染事件,为手足口病的临床诊断提供了一种精准检测方法。严重急性呼吸道感染(Severe Acute Respiratory Infection, SARI)是儿童住院和感染死亡的重要原因之一。王延群等人对135份SARI儿童和15份非SARI儿童的呼吸道样品进行了mNGS测序,检测出病毒种类和主要病毒性病原,同时也通过比较病毒基因组学分析,发现感染组和对照组之间不同的病毒群落分布特点以及各个组内的特异性病毒特征。
1、微生物组分析的一个未来临床应用可能是艰难梭菌相关疾病的管理和治疗。艰难梭菌是一种机会性细菌,可感染肠道,导致产生毒素,可导致腹泻、脱水、败血症和死亡。艰难梭菌感染只发生在受广谱抗生素或胃肠道手术等因素影响而改变的微生物组环境中。粪便移植在治疗可以治愈80% - 90%艰难梭菌感染中,这也证明了微生物组的重要性。在多项研究中使用mNGS来表征微生物组,促进了细菌-益生菌混合物的开发,这些混合物可作为预防或治疗艰难梭菌相关疾病的药片使用。
2、微生物组的另一个潜在应用是分析细菌多样性,这可以为患者的疾病是传染性还是非传染性提供线索。例如,一项用于鉴定肺炎患者呼吸道病原体的mNGS研究发现,经培养证实感染的个体其呼吸道微生物组的多样性明显较低。微生物组的改变,称为生态失调,它通常与肥胖、糖尿病、炎症性肠病等疾病相关,而微生物组的调控可能是治疗这些病理条件的途径。
微生物组分析可以告知疾病、预后在急性和慢性疾病状态。彩色条代表微生物群的类别。生物失调(一种不健康的状态)可以看到物种多样性的减少,例如难辨梭菌相关疾病患者的情况。健康个体的粪便可以通过粪便移植或口服包囊粪丸来治疗艰难梭菌感染患者。另外,健康个体微生物群产生的合成粪便也可以作为益生菌用于治疗患者。除了艰难梭菌感染,慢性疾病,如肥胖,炎症性肠病和糖尿病是益生菌治疗的潜在靶点
临床mNGS主要关注微生物reads,然而RNA病毒检测的mNGS测序恰巧可以产生宿主RNA-seq分析数据,研究人类宿主感染时的基因表达对病菌感染具有补充作用。
尽管迄今为止没有任何基于RNA-seq的检测方法被临床验证用于患者,但RNA序列分析的潜在价值非常高。通过对微生物中具有转录活性的RNA序列分析,可以区分感染与定殖、活菌与死菌。此外,对人类宿主的RNAseq分析可用于直接从临床样本中识别新的或未得到充分重视的宿主微生物相互作用,正如莱姆病、登革热或疟疾患者中所显示的那样。RNA-seq分析可能在未来临床应用中具有特别重要的作用,因为真正起作用的病原菌在转录组中是短暂存在的。与血清学检测类似,感染的间接诊断也可能基于病原体特异性的人类宿主反应。。随着大规模测序数据的不断产生,或许是由常规临床mNGS检测驱动的,对人类reads的二次挖掘可能通过整合微生物和宿主基因表达数据提高临床诊断的准确性。
在肿瘤领域,全基因组测序或Panel捕获的NGS方法可用于同时发现与癌症相关的病毒(即疱疹病毒、乳头瘤病毒和多瘤病毒)或收集病毒与宿主相互作用的数据。例如,mNGS在发现默克尔细胞多瘤病毒(图1D)中起关键作用,默克尔细胞癌是一种最常见于老年患者的罕见皮肤癌,默克尔细胞多瘤病毒现在认为是默克尔细胞癌的原因。迄今为止,美国食品和药物管理局(FDA)已经批准了两种NGS Panel(MSK,Foundation One)检测基因突变。 通过在Panel上添加特定的病毒探针 ,可以对整个肿瘤基因组或外显子组进行测序时,顺便完成这些样本中整合病毒和外源性病毒的检测。
[1] Chiu C Y , Miller S A . Clinical metagenomics[J]. Nature Reviews Genetics, 2019.